MacroAPE 1083:MSX2 f1
From FANTOM5_SSTAR
Full Name: MSX2_f1
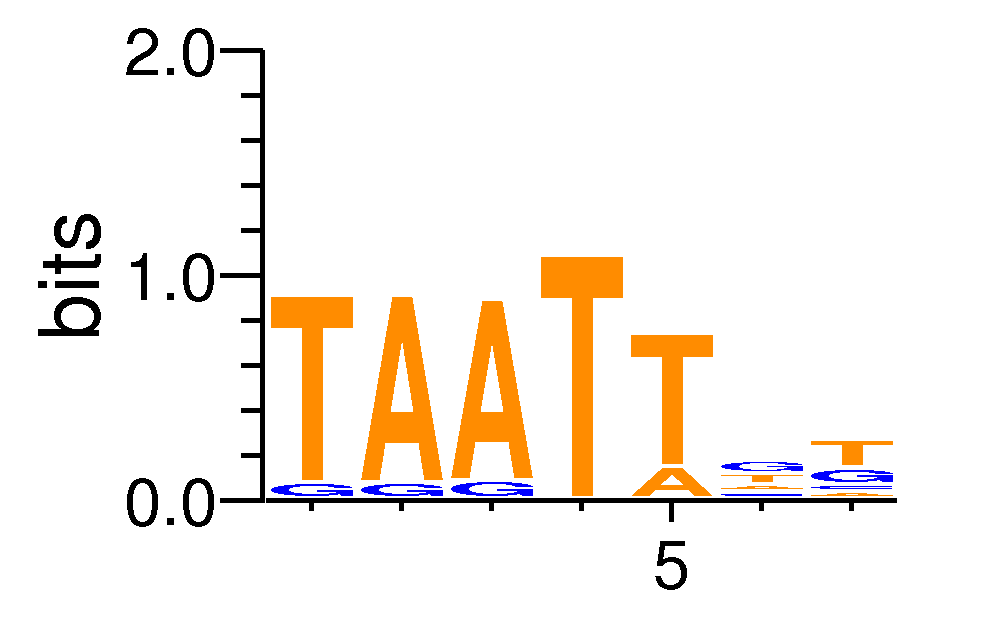
| Motif matrix | ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
Sample specificity
The values shown are the p-values of the motif in FANTOM5 samples. <br>Analyst: Michiel de Hoon
TomTom analysis
<br>Analyst: Michiel de Hoon and Hiroko Ohmiya
Target_ID | Optimal_offset | p-value | E-value | q-value | Overlap | Query consensus | Target consensus | Orientation |
|---|---|---|---|---|---|---|---|---|
| Prrx2#MA0075.1 | 0 | 5.04198e-05 | 0.0239998 | 0.0401071 | 5 | TAATTGT | TAATT | - |
| Dll#MA0187.1 | 0 | 0.000115401 | 0.0549309 | 0.0401071 | 7 | TAATTGT | TAATTAC | + |
| YHP1#MA0426.1 | 0 | 0.000306061 | 0.145685 | 0.0401071 | 6 | TAATTGT | TAATTG | + |
| HAT5#MA0008.1 | 2 | 0.000339233 | 0.161475 | 0.0401071 | 6 | TAATTGT | AATAATTG | - |
| Nobox#MA0125.1 | 0 | 0.000408912 | 0.194642 | 0.0401071 | 7 | TAATTGT | TAATTGGT | + |
| PHDP#MA0457.1 | 1 | 0.00044635 | 0.212462 | 0.0401071 | 6 | TAATTGT | TTAATTA | + |
| Dr#MA0188.1 | 0 | 0.000484251 | 0.230503 | 0.0401071 | 7 | TAATTGT | TAATTGG | - |
| unc-4#MA0250.1 | 1 | 0.000484251 | 0.230503 | 0.0401071 | 6 | TAATTGT | TTAATTG | + |
| CG13424#MA0175.1 | 1 | 0.000611235 | 0.290948 | 0.0401071 | 6 | TAATTGT | TTAATTG | + |
| CG32532#MA0179.1 | 1 | 0.000708643 | 0.337314 | 0.0401071 | 6 | TAATTGT | TTAATTA | + |
| hbn#MA0226.1 | 1 | 0.000708643 | 0.337314 | 0.0401071 | 6 | TAATTGT | TTAATTA | + |
| CG34031#MA0444.1 | 1 | 0.000708643 | 0.337314 | 0.0401071 | 6 | TAATTGT | TTAATTG | + |
| Hmx#MA0192.1 | 1 | 0.000761705 | 0.362571 | 0.0401071 | 6 | TAATTGT | TTAATTG | + |
| NK7.1#MA0196.1 | 1 | 0.000761705 | 0.362571 | 0.0401071 | 6 | TAATTGT | TTAATTG | + |
| CG11085#MA0171.1 | 1 | 0.000878674 | 0.418249 | 0.0401071 | 6 | TAATTGT | TTAATTG | + |
| Ubx#MA0094.2 | 0 | 0.000934536 | 0.444839 | 0.0401071 | 4 | TAATTGT | TAAT | + |
| OdsH#MA0455.1 | 1 | 0.000943413 | 0.449065 | 0.0401071 | 6 | TAATTGT | CTAATTA | + |
| en#MA0220.1 | 1 | 0.000943413 | 0.449065 | 0.0401071 | 6 | TAATTGT | TTAATTA | + |
| slou#MA0245.1 | 1 | 0.000943413 | 0.449065 | 0.0401071 | 6 | TAATTGT | TTAATTA | + |
| OdsH#MA0455.1 | 1 | 0.000943413 | 0.449065 | 0.0401071 | 6 | TAATTGT | CTAATTA | + |
| CG15696#MA0176.1 | 1 | 0.00101349 | 0.482423 | 0.0410347 | 6 | TAATTGT | TTAATTA | + |
| Nkx2-5#MA0063.1 | 1 | 0.00108595 | 0.51691 | 0.0412657 | 6 | TAATTGT | TTAATTG | + |
| B-H2#MA0169.1 | 1 | 0.0011648 | 0.554444 | 0.0412657 | 6 | TAATTGT | TTAATTG | + |
| Pph13#MA0200.1 | 1 | 0.0011648 | 0.554444 | 0.0412657 | 6 | TAATTGT | CTAATTA | + |
| Rx#MA0202.1 | 1 | 0.00125745 | 0.598547 | 0.0427662 | 6 | TAATTGT | CTAATTA | + |
| Pdx1#MA0132.1 | 1 | 0.00139349 | 0.6633 | 0.0435283 | 5 | TAATTGT | CTAATT | + |
| ATHB-5#MA0110.1 | 2 | 0.00153171 | 0.729094 | 0.0435283 | 7 | TAATTGT | AATAATTGG | - |
| Vsx1#MA0181.1 | 1 | 0.00153583 | 0.731054 | 0.0435283 | 6 | TAATTGT | TTAATTA | + |
| unpg#MA0251.1 | 1 | 0.00153583 | 0.731054 | 0.0435283 | 6 | TAATTGT | TTAATTA | + |
| YOX1#MA0433.1 | 1 | 0.00172042 | 0.818922 | 0.0471872 | 7 | TAATTGT | TTAATTAA | + |
| CG9876#MA0184.1 | 1 | 0.00214405 | 1.02057 | 0.0569686 | 6 | TAATTGT | CTAATTA | + |
| Lim3#MA0195.1 | 0 | 0.00247902 | 1.18002 | 0.0619943 | 7 | TAATTGT | TAATTAA | - |
| repo#MA0240.1 | 1 | 0.00247902 | 1.18002 | 0.0619943 | 6 | TAATTGT | TTAATTA | + |
| tup#MA0248.1 | 1 | 0.00265534 | 1.26394 | 0.0645063 | 6 | TAATTGT | TTAATTG | + |
| inv#MA0229.1 | 2 | 0.00288508 | 1.3733 | 0.0668756 | 6 | TAATTGT | TCTAATTA | + |
| lbl#MA0232.1 | 0 | 0.00291018 | 1.38524 | 0.0668756 | 6 | TAATTGT | TAATTA | + |
| otp#MA0236.1 | 1 | 0.00302441 | 1.43962 | 0.0676718 | 6 | TAATTGT | TTAATTA | + |
| CG32105#MA0178.1 | 1 | 0.00321752 | 1.53154 | 0.0701466 | 6 | TAATTGT | TTAATTA | + |
| bsh#MA0214.1 | 1 | 0.00341737 | 1.62667 | 0.0726411 | 6 | TAATTGT | TTAATTG | + |
| HGTX#MA0191.1 | 1 | 0.00362636 | 1.72615 | 0.0730826 | 6 | TAATTGT | TTAATTA | + |
| ro#MA0241.1 | 1 | 0.00362636 | 1.72615 | 0.0730826 | 6 | TAATTGT | CTAATTA | + |
| SMP1#MA0383.1 | 8 | 0.003696 | 1.7593 | 0.0730826 | 7 | TAATTGT | TGCTAATTTAATTATAGGTAA | - |
| B-H1#MA0168.1 | 1 | 0.00407127 | 1.93792 | 0.0752527 | 6 | TAATTGT | TTAATTG | + |
| exex#MA0224.1 | 1 | 0.00407127 | 1.93792 | 0.0752527 | 6 | TAATTGT | GTAATTA | + |
| exex#MA0224.1 | 1 | 0.00407127 | 1.93792 | 0.0752527 | 6 | TAATTGT | GTAATTA | + |
| Awh#MA0167.1 | 1 | 0.00481527 | 2.29207 | 0.0818843 | 6 | TAATTGT | TTAATTA | + |
| C15#MA0170.1 | 1 | 0.00481527 | 2.29207 | 0.0818843 | 6 | TAATTGT | TTAATTA | + |
| Lhx3#MA0135.1 | 7 | 0.0049362 | 2.34963 | 0.0822948 | 6 | TAATTGT | GATTAATTAATTT | - |
| Ubx#MA0094.2 | 2 | 0.00509224 | 2.4239 | 0.0832636 | 6 | TAATTGT | TTTAATTA | + |
| CG18599#MA0177.1 | 0 | 0.00565934 | 2.69385 | 0.0859266 | 7 | TAATTGT | TAATTAA | - |
| Lim1#MA0194.1 | 1 | 0.00565934 | 2.69385 | 0.0859266 | 6 | TAATTGT | TTAATTA | + |
| ap#MA0209.1 | 1 | 0.00565934 | 2.69385 | 0.0859266 | 6 | TAATTGT | CTAATTA | + |
| Oct#MA0197.1 | 2 | 0.0060091 | 2.86033 | 0.0865979 | 6 | TAATTGT | TTTAATTA | + |
| E5#MA0189.1 | 1 | 0.00628929 | 2.9937 | 0.0876641 | 6 | TAATTGT | TTAATTA | + |
| Vsx2#MA0180.1 | 2 | 0.00640269 | 3.04768 | 0.0878052 | 7 | TAATTGT | TTTAATTAG | + |
| abd-A#MA0206.1 | 1 | 0.00662932 | 3.15556 | 0.0894702 | 6 | TAATTGT | TTAATTA | + |
| CG11294#MA0172.1 | 1 | 0.00698197 | 3.32342 | 0.0913302 | 6 | TAATTGT | TTAATTA | + |
| al#MA0208.1 | 1 | 0.00698197 | 3.32342 | 0.0913302 | 6 | TAATTGT | TTAATTA | - |
| zen2#MA0257.1 | 1 | 0.00735096 | 3.49906 | 0.0942111 | 6 | TAATTGT | TTAATTA | + |
| CG7056#MA0183.1 | 0 | 0.0074246 | 3.53411 | 0.0942111 | 7 | TAATTGT | TAATTAAA | - |
| MATALPHA2#MA0328.1 | 4 | 0.00753461 | 3.58648 | 0.0942111 | 5 | TAATTGT | CATGTAATT | + |
| HNF1A#MA0046.1 | 3 | 0.0081001 | 3.85565 | 0.0954252 | 7 | TAATTGT | GGTTAATAATTAAC | + |
| SUM1#MA0398.1 | 2 | 0.00836952 | 3.98389 | 0.0954252 | 7 | TAATTGT | AAAAATTTT | - |
| ind#MA0228.1 | 1 | 0.00897848 | 4.27376 | 0.0954252 | 6 | TAATTGT | CTAATTA | + |
| CG4328#MA0182.1 | 1 | 0.00942791 | 4.48768 | 0.0977578 | 6 | TAATTGT | TTTATTA | + |
| ftz#MA0225.1 | 1 | 0.0098963 | 4.71064 | 0.0989929 | 6 | TAATTGT | TTAATGA | + |
| ems#MA0219.1 | 1 | 0.0103833 | 4.94245 | 0.101477 | 6 | TAATTGT | TTAATGA | + |
| PHO2#MA0356.1 | 1 | 0.0111033 | 5.28515 | 0.103782 | 5 | TAATTGT | ATAATA | + |
| lab#MA0230.1 | 1 | 0.0114148 | 5.43343 | 0.103782 | 6 | TAATTGT | TTAATTA | + |
| pb#MA0238.1 | 0 | 0.0114148 | 5.43343 | 0.103782 | 7 | TAATTGT | TAATTAA | - |
| HOXA5#MA0158.1 | 3 | 0.0122259 | 5.81953 | 0.105001 | 5 | TAATTGT | CACTAATT | + |
| H2.0#MA0448.1 | 1 | 0.0125313 | 5.96491 | 0.105493 | 6 | TAATTGT | TTAATTA | + |
| eve#MA0221.1 | 1 | 0.0143797 | 6.84475 | 0.116355 | 6 | TAATTGT | CTAATGA | + |
| lbe#MA0231.1 | 0 | 0.0145057 | 6.90473 | 0.116355 | 6 | TAATTGT | TAACTA | + |