MacroAPE 1083:ISL1 f1
From FANTOM5_SSTAR
Full Name: ISL1_f1
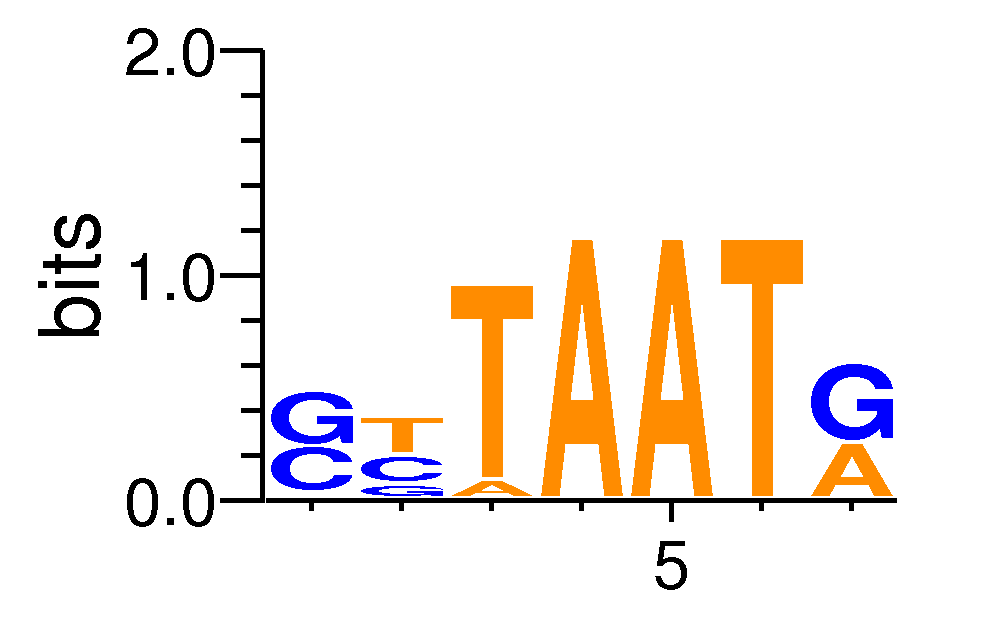
| ExpandMotif matrix |
|---|
Sample specificity
The values shown are the p-values of the motif in FANTOM5 samples. <br>Analyst: Michiel de Hoon
TomTom analysis
<br>Analyst: Michiel de Hoon and Hiroko Ohmiya
Target_ID | Optimal_offset | p-value | E-value | q-value | Overlap | Query consensus | Target consensus | Orientation |
|---|---|---|---|---|---|---|---|---|
| Dfd#MA0186.1 | -1 | 0.00022717 | 0.108133 | 0.133871 | 6 | GTTAATG | TTAATGA | + |
| Scr#MA0203.1 | -1 | 0.000431527 | 0.205407 | 0.133871 | 6 | GTTAATG | TTAATGA | + |
| zen#MA0256.1 | -1 | 0.000758685 | 0.361134 | 0.133871 | 6 | GTTAATG | CTAATGA | + |
| HNF1B#MA0153.1 | -1 | 0.000826101 | 0.393224 | 0.133871 | 6 | GTTAATG | TTAATATTTAAC | + |
| Ubx#MA0094.2 | -2 | 0.000902999 | 0.429828 | 0.133871 | 4 | GTTAATG | TAAT | + |
| E5#MA0189.1 | -1 | 0.00116646 | 0.555234 | 0.133871 | 6 | GTTAATG | TTAATTA | + |
| ems#MA0219.1 | -1 | 0.00116646 | 0.555234 | 0.133871 | 6 | GTTAATG | TTAATGA | + |
| eve#MA0221.1 | -1 | 0.00116646 | 0.555234 | 0.133871 | 6 | GTTAATG | CTAATGA | + |
| btn#MA0215.1 | -1 | 0.00142546 | 0.678518 | 0.145418 | 6 | GTTAATG | TTAATGA | + |
| pb#MA0238.1 | -1 | 0.00225379 | 1.07281 | 0.176572 | 6 | GTTAATG | TTAATTA | + |
| Antp#MA0166.1 | -1 | 0.00246212 | 1.17197 | 0.176572 | 6 | GTTAATG | TTAATGA | + |
| bsh#MA0214.1 | -1 | 0.00246212 | 1.17197 | 0.176572 | 6 | GTTAATG | TTAATTG | + |
| HNF1A#MA0046.1 | 1 | 0.00250011 | 1.19005 | 0.176572 | 7 | GTTAATG | GGTTAATAATTAAC | + |
| ftz#MA0225.1 | -1 | 0.00292267 | 1.39119 | 0.178894 | 6 | GTTAATG | TTAATGA | + |
| zen2#MA0257.1 | -1 | 0.00292267 | 1.39119 | 0.178894 | 6 | GTTAATG | TTAATTA | + |
| tup#MA0248.1 | -1 | 0.00321423 | 1.52997 | 0.184444 | 6 | GTTAATG | TTAATTG | + |
| ind#MA0228.1 | -1 | 0.00410513 | 1.95404 | 0.221709 | 6 | GTTAATG | CTAATTA | + |
| Pdx1#MA0132.1 | -1 | 0.00494095 | 2.35189 | 0.239867 | 6 | GTTAATG | CTAATT | + |
| CG13424#MA0175.1 | -1 | 0.00548635 | 2.6115 | 0.239867 | 6 | GTTAATG | TTAATTG | + |
| CG18599#MA0177.1 | -1 | 0.00548635 | 2.6115 | 0.239867 | 6 | GTTAATG | CTAATTA | + |
| lab#MA0230.1 | -1 | 0.00548635 | 2.6115 | 0.239867 | 6 | GTTAATG | TTAATTA | + |
| slou#MA0245.1 | -1 | 0.00627149 | 2.98523 | 0.252711 | 6 | GTTAATG | TTAATTA | + |
| Oct#MA0197.1 | 0 | 0.00633061 | 3.01337 | 0.252711 | 7 | GTTAATG | TTTAATTA | + |
| ap#MA0209.1 | -1 | 0.00669132 | 3.18507 | 0.255539 | 6 | GTTAATG | CTAATTA | + |
| HGTX#MA0191.1 | -1 | 0.00759117 | 3.6134 | 0.255539 | 6 | GTTAATG | TTAATTA | + |
| CG11085#MA0171.1 | -1 | 0.00807139 | 3.84198 | 0.255539 | 6 | GTTAATG | TTAATTG | + |
| NK7.1#MA0196.1 | -1 | 0.00807139 | 3.84198 | 0.255539 | 6 | GTTAATG | TTAATTG | + |
| abd-A#MA0206.1 | -1 | 0.00807139 | 3.84198 | 0.255539 | 6 | GTTAATG | TTAATTA | + |
| H2.0#MA0448.1 | -1 | 0.00807139 | 3.84198 | 0.255539 | 6 | GTTAATG | TTAATTA | + |
| Vsx1#MA0181.1 | -1 | 0.00910777 | 4.3353 | 0.269747 | 6 | GTTAATG | TTAATTA | + |
| otp#MA0236.1 | -1 | 0.00910777 | 4.3353 | 0.269747 | 6 | GTTAATG | TTAATTA | + |
| Lim3#MA0195.1 | -1 | 0.0096673 | 4.60164 | 0.277371 | 6 | GTTAATG | TTAATTA | + |
| Awh#MA0167.1 | -1 | 0.0122838 | 5.84711 | 0.341764 | 6 | GTTAATG | TTAATTA | + |
| lbl#MA0232.1 | -2 | 0.0143761 | 6.84301 | 0.353516 | 5 | GTTAATG | TAATTA | + |
| C15#MA0170.1 | -1 | 0.0145236 | 6.91322 | 0.353516 | 6 | GTTAATG | TTAATTA | + |
| unpg#MA0251.1 | -1 | 0.0145236 | 6.91322 | 0.353516 | 6 | GTTAATG | TTAATTA | + |
| en#MA0220.1 | -1 | 0.015332 | 7.29802 | 0.353516 | 6 | GTTAATG | TTAATTA | + |
| ro#MA0241.1 | -1 | 0.015332 | 7.29802 | 0.353516 | 6 | GTTAATG | CTAATTA | + |
| HOXA5#MA0158.1 | 1 | 0.0157901 | 7.5161 | 0.353516 | 7 | GTTAATG | CACTAATT | + |
| CG32532#MA0179.1 | -1 | 0.0161716 | 7.69767 | 0.353516 | 6 | GTTAATG | TTAATTA | + |
| CG34031#MA0444.1 | -1 | 0.0161716 | 7.69767 | 0.353516 | 6 | GTTAATG | TTAATTG | + |
| Ubx#MA0094.2 | 0 | 0.0186075 | 8.85717 | 0.397307 | 7 | GTTAATG | TTTAATTA | + |
| SKO1#MA0382.1 | 1 | 0.0206863 | 9.84668 | 0.409225 | 7 | GTTAATG | ACGTAATG | + |
| CG9876#MA0184.1 | -1 | 0.0209485 | 9.97149 | 0.409225 | 6 | GTTAATG | CTAATTA | + |
| unc-4#MA0250.1 | -1 | 0.0209485 | 9.97149 | 0.409225 | 6 | GTTAATG | TTAATTG | + |
| PHDP#MA0457.1 | -1 | 0.0209485 | 9.97149 | 0.409225 | 6 | GTTAATG | TTAATTA | + |