MacroAPE 1083:MAFA f1
From FANTOM5_SSTAR
Full Name: MAFA_f1
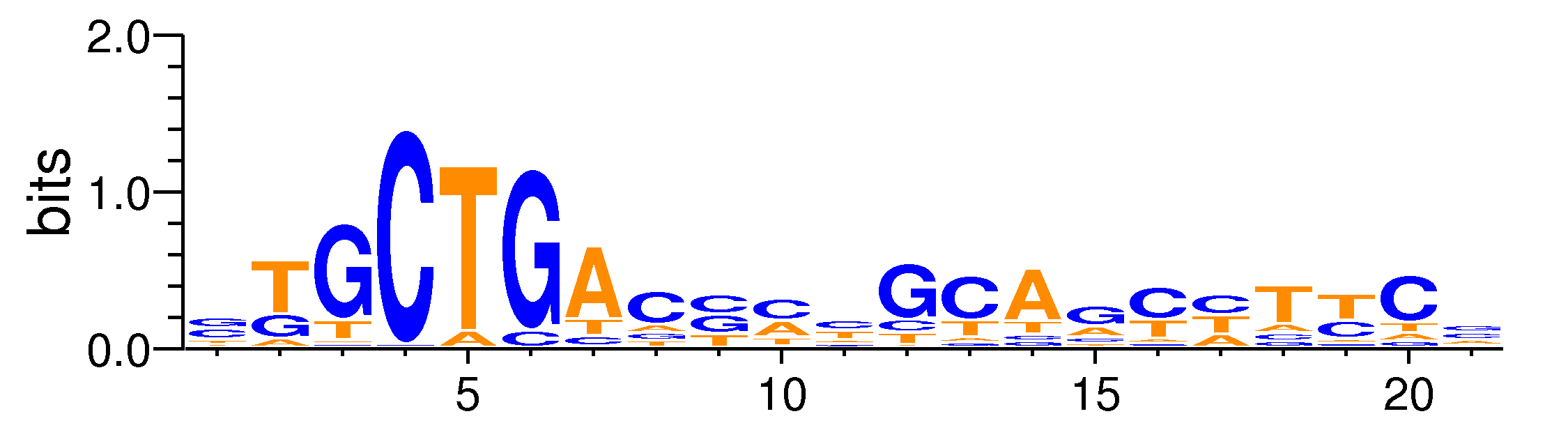
| ExpandMotif matrix |
|---|
Sample specificity
The values shown are the p-values of the motif in FANTOM5 samples. <br>Analyst: Michiel de Hoon
TomTom analysis
<br>Analyst: Michiel de Hoon and Hiroko Ohmiya
Target_ID | Optimal_offset | p-value | E-value | q-value | Overlap | Query consensus | Target consensus | Orientation |
|---|---|---|---|---|---|---|---|---|
| Mafb#MA0117.1 | -2 | 5.44193e-05 | 0.0259036 | 0.0518072 | 8 | GTGCTGACCCCGCAGCCTTCG | GCTGACGG | + |
| Pax4#MA0068.1 | 6 | 0.0019886 | 0.946576 | 0.638351 | 21 | GTGCTGACCCCGCAGCCTTCG | GAAAAATTTCCCATACTCCACTCCCCCCCC | + |
| NFE2L2#MA0150.1 | -3 | 0.00201161 | 0.957527 | 0.638351 | 11 | GTGCTGACCCCGCAGCCTTCG | ATGACTCAGCA | + |
| STP2#MA0395.1 | 0 | 0.00468114 | 2.22822 | 0.889661 | 20 | GTGCTGACCCCGCAGCCTTCG | TGATCGGCGCCGCACGACGA | + |
| YAP6#MA0418.1 | 0 | 0.00533374 | 2.53886 | 0.889661 | 20 | GTGCTGACCCCGCAGCCTTCG | GGTCTGATTACGTAAGCGAC | + |
| CREB1#MA0018.1 | 1 | 0.00604606 | 2.87793 | 0.889661 | 11 | GTGCTGACCCCGCAGCCTTCG | CCTGGTGACGTC | + |
| Pax5#MA0014.1 | -1 | 0.00816642 | 3.88722 | 0.889661 | 20 | GTGCTGACCCCGCAGCCTTCG | CCGCTACGCTTCAGTGCTCT | - |
| ACE2#MA0267.1 | -1 | 0.00841066 | 4.00347 | 0.889661 | 7 | GTGCTGACCCCGCAGCCTTCG | TGCTGGT | - |
| PHD1#MA0355.1 | -7 | 0.0150615 | 7.16928 | 1 | 10 | GTGCTGACCCCGCAGCCTTCG | ACCTGCAGCA | + |
| ESR1#MA0112.2 | 5 | 0.0176628 | 8.40748 | 1 | 15 | GTGCTGACCCCGCAGCCTTCG | AGGTCAGGGTGACCTGGGCC | - |
| USV1#MA0413.1 | 0 | 0.0176628 | 8.40748 | 1 | 20 | GTGCTGACCCCGCAGCCTTCG | AAATTCCCCCTGAATTTGTG | + |
| Trl#MA0205.1 | 0 | 0.0208446 | 9.92202 | 1 | 10 | GTGCTGACCCCGCAGCCTTCG | TTGCTCTCTC | + |